研究方向
药物新靶点的发现与功能确证是原创药物研发的核心瓶颈。代谢异常在肿瘤、免疫性疾病、心力衰竭等多种重大疾病中发挥着决定性作用。近年来的研究表明,肿瘤微环境(TME)中癌细胞、免疫细胞、神经元等多类型细胞之间的复杂代谢互作网络,在调控肿瘤进展及治疗响应中具有关键作用,成为当前研究的重要焦点。尤其值得关注的是,神经系统对于肿瘤进展的调控作用正日益凸显。“肿瘤—神经—免疫”轴介导的代谢互作机制,正在成为解析复杂病理机制和识别靶向干预通路的重要突破口。
本实验室聚焦肿瘤等疾病中的代谢重塑机制与药物靶点发现,研发新型单细胞代谢组学与AI驱动的多组学整合分析技术,结合生物信息学分析与AI算法模型,利用细胞、分子和代谢生物学等方法,系统解析细胞内及组织微环境中的代谢异质性与跨细胞代谢互作网络,深入探究其功能与调控机制,从而提供新型治疗策略和潜在新药靶点。核心研究方向包括:
1. TME中“肿瘤—神经—免疫”轴的多细胞代谢互作与单细胞代谢异质性
结合微量代谢组学与单细胞多组学技术,系统解析TME中不同类型细胞(如癌细胞、免疫细胞、神经元等)之间的代谢互作网络,揭示代谢重塑的单细胞水平时空异质性及其在疾病进展中的生物学功能与调控机制。重点关注神经系统介导的“肿瘤—神经—免疫”代谢轴,阐明其在肿瘤进展与治疗响应中的关键作用,揭示潜在干预通路与治疗靶点。
2. 抗癌药物耐受性(DTP)的形成与进展中的代谢重塑与调控机制
揭示肿瘤治疗drug tolerant persister(DTP)细胞的形成、维持与进化过程中的代谢重塑与适应机制,探索其与TME中神经、免疫等各类细胞间的代谢互作,揭示驱动DTP的关键调控因子与潜在干预策略。
3. 前沿跨尺度代谢组学与AI驱动的多组学整合技术开发
自主研发适用于痕量样本和单细胞的高灵敏、宽覆盖、高通量、高保真的代谢组学与代谢流分析技术,以及AI驱动的多组学数据整合与智能化分析方法,赋能从分子机制建模到靶点识别的系统性研究。
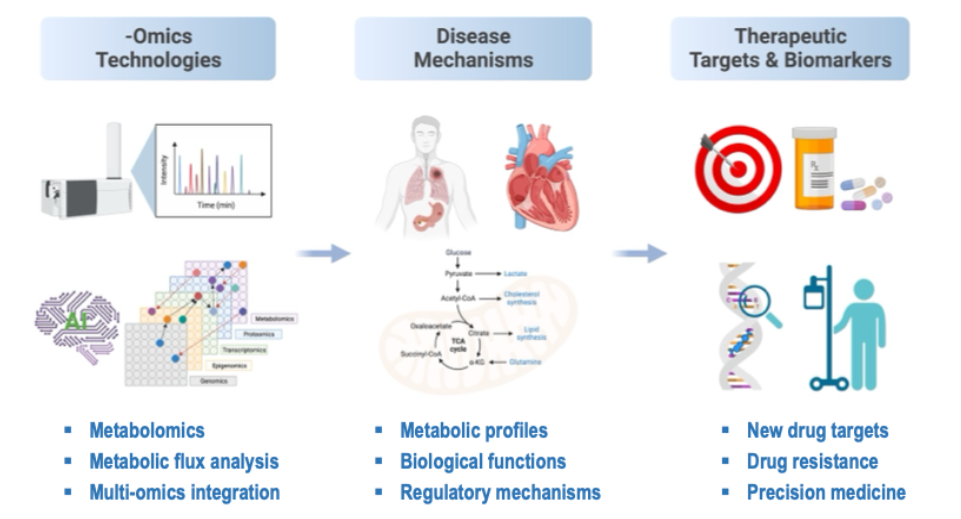
胡泽平课题组研究方向概览
研究成果
1. 研发AI驱动的跨尺度、高灵敏代谢组学与多组学整合分析技术
研发基于质谱平台和AI的微量、单细胞等跨尺度代谢组学分析技术,实现对极微样本量(如早期胚胎、少量细胞)的代谢动态精准解析;结合新型衍生化策略与代谢流示踪技术,推动从组织至细胞水平的代谢测量能力跃升,为发育、干细胞和肿瘤等疾病模型中的微环境代谢研究提供关键技术工具。
2. 解析肿瘤等重大疾病的代谢重塑规律与关键调控机制
系统研究肿瘤(如肺癌、胰腺癌、乳腺癌)和心血管疾病的代谢网络重构与细胞互作机制,揭示其在肿瘤分型、转移、耐药及心衰等过程中的关键作用,鉴定靶向嘌呤、乙酰胆碱、胆汁酸等代谢通路的新型治疗靶点和预测标志物,并扩展至病毒感染相关疾病的代谢病理机制,为疾病诊断与干预提供理论支撑和临床策略依据。
3. 新药靶点发现与转化医学
以代谢组学为核心,联合AI与多组学整合,开展靶点发现、疾病分型与精准预测研究,成功应用于早期肺腺癌、胃癌和心肌病等疾病模型;相关研究揭示多个可成药代谢通路,部分验证已有药物的再定位价值,建立起从代谢机制解析到靶点验证和干预策略设计的完整研究链条。
荣誉和奖项
- 2017年 拜耳学者奖(Bayer Investigator Award)
- 2019年 第十四届全国医药卫生青年科技论坛二等奖
- 2019年 年度优秀委员,中国抗癌协会肿瘤代谢专委会
- 2019年 优秀教学奖,清华大学药学院
- 2020年 拜耳学者奖(Bayer Investigator Award)
- 2022年 清华大学优秀博士论文导师
- 2022年 教育部“长江学者”特聘教授
代表性论文(按时间倒序,*通讯作者;#第一作者)
研究论文
1. Chen Y#, Wang B#, Zhao Y#, Shao X#, Wang M#, Ma F#, Yang L, Nie M, Jin P, Yao K, Song H, Lou S, Wang H, Yang T, Tian Y*, Han P*, Hu ZP*. Metabolomics machine learning predictor of diagnosis and prognosis of gastric cancer. Nature Communications. 2024;15(1):1657.
2. Fan H#, Xu Z#, Yao K#, Zheng B, Zhang Y, Wang X, Zhang T, Li X, Hu H, Yue B*, Hu ZP*, Zheng H*. Osteoclast cancer cell metabolic cross-talk confers PARP inhibitor resistance in bone metastatic breast cancer. Cancer Research. 2024;84(3):449-467.
3. Nie M#, Chen N#, Pang H#, Jiang T#, Jiang W, Tian P, Yao L, Chen Y, DeBerardinis RJ, Li W, Yu Q, Zhou C, Hu ZP*. Targeting acetylcholine signaling modulates persistent drug tolerance in EGFR-mutant lung cancer and impedes tumor relapse. Journal of Clinical Investigation. 2022;132(20):e160152.
4. Yuan M#, Tu B#, Li H#, Pang H#, Zhang N, Bai J, Shu Z, Christopher C, Huo S, Zhai J, Yao K, Wang L, Ying H, Zhu WG, Fu D, Hu ZP*, Zhao Y*. Cancer associated fibroblasts employ NUFIP1-dependent autophagy to secrete nucleosides and support pancreatic tumor growth. Nature Cancer. 2022;3(8):945-960.
5. Wang W#, Wang J#,*, Yao K#, Wang S#, Nie M, Xu J, Wu G, Lu M, Pei H, Luo X, Li D, Yang T, Li P, Song L*, Hu ZP*. Metabolic characterization of hypertrophic cardiomyopathy in human hearts. Nature Cardiovascular Research. 2022;1:445–461.
6. Jiang Y*,#, Deng Y#, Pang H#, Ma T, Ye Q, Chen Q, Chen H, Hu ZP*, Qin CF*, Xu Z*. Treatment of SARS-CoV-2 induced pneumonia with NAD+ and NMN in two mouse models. Cell Discovery. 2022;8(1):38.
7. Cheng W#, Pang H#, Campen MJ, Zhang J, Li Y, Gao J, Ren D, Ji X, Rothman N, Lan Q, Zheng Y, Leng S*, Hu ZP*, Tang J*. Circulatory metabolites trigger ex vivo arterial endothelial cell dysfunction in population chronically. Particle and Fibre Toxicology. 2022;19(1):20.
8. Nie M#, Yao K#, Zhu X#, Chen N, Xiao N, Wang Y, Peng B, Yao LA, Li P, Zhang P*, Hu ZP*. Evolutionary metabolic landscape from preneoplasia to invasive lung adenocarcinoma with therapeutic implications. Nature Communications. 2021;12(1):6479.
9. Zhao J#, Yao K#, Yu H#, Zhang L#, Xu Y#, Chen L, Sun Z, Zhu Y, Zhang C, Qian Y, Ji S, Pang H, Zhang M, Chen J, Correia C, Weiskittel T, Lin DW, Zhao Y, Chandrasekaran S, Fu X, Zhang D, Fan HY, Xie W, Li H, Hu ZP*, Zhang J*. Metabolic remodeling during early murine embryo development. Nature Metabolism. 2021;3(10):1372-1384.
10. Pang H#, Jiang Y#, Li J#, Wang Y#, Nie M#, Xiao N, Wang S, Song Z, Ji F, Chang Y, Zheng Y, Yao K, Yao L, Li S, Song L*, Lan X*, Xu Z*, Hu ZP*. Aberrant NAD+ metabolism underlies ZIKA virus-induced microcephaly. Nature Metabolism. 2021;3(8):1109-1124.
11. Xiao N#, Nie M#, Pang H#, Wang B#, Hu J#, Meng X, Li K, Ran X, Long Q, Deng H, Chen N, Li S, Tang N*, Huang A*, Hu ZP*. Integrated cytokine and metabolite analysis reveals immunometabolic reprogramming in COVID-19 patients with therapeutic implications. Nature Communications. 2021;12(1):1618.
12. Meng X#, Pang H#, Sun F, Jin X, Wang B, Yao K, Yao L, Wang L, Hu ZP*. Simultaneous 3-nitrophenylhydrazine derivatization strategy of carbonyl, carboxyl and phosphoryl submetabolome for LC-MS/MS-based targeted metabolomics with improved sensitivity and coverage. Analytical Chemistry. 2021;93(29):10075-10083.
13. Shi Z#, Xu S#, Xing S, Yao K, Zhang L, Xue L, Zhou P, Wang M, Yan G, Yang P, Liu J, Hu ZP*, Lan F*. Mettl17, a regulator of mitochondrial ribosomal RNA modifications, is required for the translation of mitochondrial coding genes. The FASEB Journal. 2019;33(11):13040-13050.
14. Huang F, Ni M, Chalishazar MD, Huffman KE, Kim J, Cai L, Shi X, Cai F, Zacharias LG, Ireland AS, Li K, Gu W, Kaushik AK, Liu X, Gazdar AF, Oliver TG, Minna JD, Hu ZP*, DeBerardinis RJ*. Inosine monophosphate dehydrogenase dependence in a subset of small cell lung cancers. Cell Metabolism. 2018;28(3):369-382.
15. Li XK#, Lu QB#, Chen WW#, Xu W#, Liu R, Zhang SF, Du J, Li H, Yao K, Zhai D, Zhang PH, Xing B, Cui N, Yang ZD, Yuan C, Zhang XA, Xu Z, Cao WC*, Hu ZP*, Liu W*. Arginine deficiency is involved in thrombocytopenia and immunosuppression in Severe Fever with Thrombocytopenia Syndrome. Science Translational Medicine. 2018;10(459): eaat4162.
综述论文
16. Xu Y, Jiang X, Hu ZP*. Synergizing metabolomics and artificial intelligence for advancing precision oncology. Trends in Molecular Medicine. 2025:S1471-4914(25)00016-4.
17. Nie M*, Hu ZP*. Metabolic orchestration of drug-tolerant persister cells in cancer. Life Medicine. 2024;3(6):lnae040.
18. Wang F#, Fu K#, Wang Y, Pan C, Wang X, Liu Z, Yang C, Zheng Y, Li X, Lu Y, To KKW, Xia C, Zhang J, Shi Z, Hu ZP*, Huang M*, Fu L*. Small-molecule agents for cancer immunotherapy. Acta Pharmaceutica Sinica B (APSB). 2024;14(3):905-952.
19. Thompson CB*, Vousden KH*, Johnson RS*, Koppenol WH*, Sies H*, Lu Z*, Finley LWS*, Frezza C*, Kim J*, Hu ZP*, Bartman CR*. A century of the Warburg effect. Nature Metabolism. 2023;5(11):1840-1843.
20. Pang H, Hu ZP*. Metabolomics in drug research and development: advances in technologies and applications. Acta Pharmaceutica Sinica B (APSB). 2023;13(8):3238-3251.
21. Wang B#, Yao K#, Hu ZP*. Advances in mass spectrometry-based single-cell metabolite analysis. TrAC Trends in Analytical Chemistry. 2023;163:117075.
22. Liang L, Sun F, Wang H, Hu ZP*. Metabolomics, metabolic flux analysis and cancer pharmacology. Pharmacology & Therapeutics. 2021;224:107827.
23. Pang H, Jia W*, Hu ZP*. Emerging applications of metabolomics in clinical pharmacology. Clinical Pharmacology & Therapeutics. 2019;106(3):544-556.

