2019年5月15日,清华大学药学院2014级本科生唐子琦在优秀本科生国际交流项目中赴UCSF交流学习期间以第一作者身份在《自然·通讯》在线发表了题为《Interpretable classification of Alzheimer’s disease pathologies with a convolutional neural network pipeline》的研究论文,首次报道了利用深度学习算法自动且有效地对阿尔兹海默症患者大脑中的β-淀粉样蛋白斑块(Amyloid-β plaque)和血管异常进行病理学分析。该研究提出了一种深度学习的训练方法,使得计算机能够精确的检测人脑组织中β-淀粉样蛋白斑块,此方法揭示了自动化阿尔兹海默症研究关键部分的可能。
阿尔兹海默症是痴呆症中最常见的一种,但对该疾病成因和治疗方面的研究进展十分缓慢。β-淀粉样蛋白斑块作为阿尔兹海默症的病理学标志之一,是神经病理学中的重要研究对象,但识别和分析大量人脑组织样本中不同形态的β-淀粉样蛋白斑块沉淀十分困难且耗时。
为了增强对阿尔兹海默症病理学的分析能力,研究人员设计了一个卷积神经网络(CNN)模型。CNN是一种深度学习算法,能够从大量数据中提取潜在的规律和模式,并据此对新的数据进行预测。为了获得足够的数据来训练CNN进行人脑组织病理学分析,研究人员开发了一个网络平台“Blob or Not”,使得病理学家能够高效的标注数以万计的潜在斑块区域图片。研究小组利用由7万张已标注的病理学图片组成的数据集来训练CNN模型,以识别阿尔兹海默症中不同类型的大脑变化,包括区分Cored plaque和Diffuse plaque,以及识别脑血管中的异常病变。研究结果表明,此算法能够以98.7%的准确率处理整个大脑切片,处理速度只受限于所使用的计算机处理器数量。同时,研究小组对此算法的可靠性和可解释性进行了严格测试,以确保其在生物学分析上是有效的。在未来,研究人员希望这些算法将成为神经病理学研究的标准部分,帮助病理学家分析大量实验和临床数据,打开精确治疗痴呆症的大门。
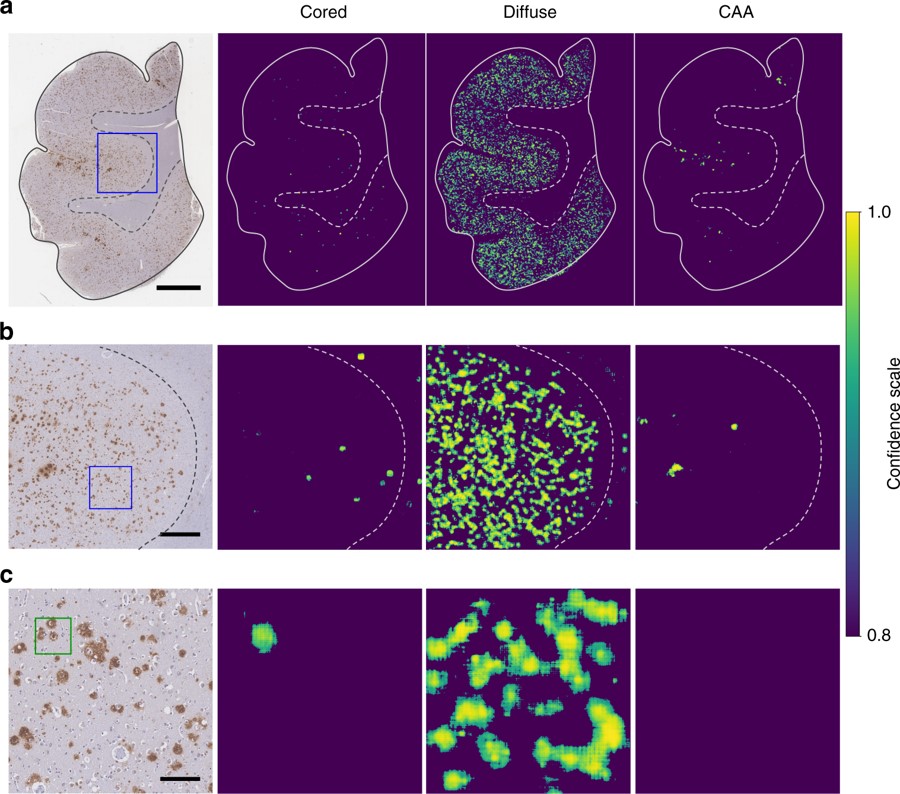
图:CNN模型在不同尺度上对人脑切片中病理学特征的预测
该工作由UCSF的Michael Keiser课题组和UC Davis的Brittany Dugger课题组合作完成,Michael Keiser教授和Brittany Dugger教授为共同通讯作者。该课题受到National Institutes of Health (P30 AG010129)和a Paul G. Allen Family Foundation Distinguished Investigator Award的资助。唐子琦同学在UCSF两年的交流学习受到中国留学基金委的资助。
原文链接:
https://doi.org/10.1038/s41467-019-10212-1


